Les tests d’égalités des fonctions de survie entre différentes valeurs d’une covariable sont calculés à partir de la méthode de Kaplan Meier.
L’utilisation du test correspond à la nécessité de déterminer si une même distribution gouverne les évènements observés dans les différentes strates.
Attention: pas de test possible sur des variables quantitatives. Il faut donc prévoir des regroupements pour les transformer en variable ordinale.
Deux méthodes sont utilisées:
La plus ancienne, la plus diffusée, et peut-être la moins bonne: test dits du log-rank).
Plus récente et (hélas) moins difusée: comparaison des RMST (Restricted Mean of Survival Time).
6.1 Tests du log-rank
Il s’agit d’une série de tests qui répondent à la même logique, la seule différence réside dans le poids accordé au début ou à la fin de la période d’observation. Par ailleurs ces différents tests sont plus ou moins sensibles à la distribution des censures à droites entre les sous échantillons et à l’hypothèses de proportionnalité des risques.
Dans leur logique, ces tests entrent dans le cadre des tests d’indépendance du Khi2, même si formellement ils relèvent des techniques dites de rang (d’où le nom).
Il s’agira donc de comparer des effectifs observés à des effectifs espérés à chaque moment d’évènement. La principale différence réside dans le calcul de la variance de la statistique du test qui, ici, suit assez logiquement une loi hypergéométrique [proche loi binomiale mais avec tirage avec remise].
6.1.1 Principe de calcul de la statistique de test
Effectifs observés en \(t_i\): \(o_{i1}\) et \(o_{i2}\) sont égaux à \(d_{i1}\) et \(d_{i2}\), et leur somme pour tous les temps d’évènement à \(O_1\) et \(O_2\).
Effectifs expérés (hypothèse nulle \(H_0\)): comme pour une statistique du \(\chi^2\) on se base sur les marges, avec le risque set (\(R_i\)) en \(t_i\) pour dénombrer les effectifs, soit \(e_{i1}=R_{i1}\times\frac{d_i}{R_i}\) et \(e_{i2}=R_{i2}\times\frac{d_2}{R_i}\). Leur somme pour tous les temps d’évènement est égale à \(E_1\) et \(E_2\). Le principe de calcul des effectifs observés reposent donc sur l’hypothèse d’un rapport des risques toujours égal à 1 au cours du temps (hypothèse fondamentale de risques proportionnels).
Statistique du log-rank: \((O_1 - E_1) = -(O_2 - E_2)\).
Statistique de test: sous \(H_0\), \(\frac{(O_1 - E_1)^2}{\sum{v_i}}\), avec \(v_i\) la variance de \((o_{i1} - e_{i2})\), suis un \(\chi^2(1)\). Si on teste simultanément la différence de \(g\) fonctions de survie, ce qui n’est pas une bonne idée en passant, la statistique de test suis un \(\chi^2(g-1)\).
6.1.2 Les principaux tests log-rank
Le principe de construction des effectifs observés et espérés reste strictement le même dans chaque test, les différences résident dans les pondérations (\(w_i\)) qui prennent en compte, de manière différente, la taille de la population encore soumise au risque à chaque durée où au moins un évènement est observé.
Test du log-rank: \(w_i=1\)
Il accorde le même poids à toutes les durées d’évènement. C’est le test standard, le plus utilisé. Il n’y a donc pas de pondération au final.
Test de Wilconxon-Breslow-Grehan: \(w_i=R_i\)
Les écarts entre effectifs observés et espérés sont pondérés par la population soumise à risque en \(t_i\). Le test accorde plus de poids au début de la période analysée, et il est sensible aux différences de distributions entre les strates des observations censurées.
Test de Tarone-Ware: \(w_i=\sqrt{R_i}\)
Variante du test précédent, il atténue le poids accordé aux évènements au début de la période d’observation. Il est par ailleurs moins sensible au problème de la distribution des censures entre les strates.
Test de Peto-Peto : \(w_i=S_i\)
La pondération est une variante de la fonction de survie KM (avec \(R_i=R_i+1\)). Le test n’est pas sensible au problème de distribution des censures.
Test de Fleming-Harington: \(w_i=(S_i)^p\times(1-S_i)^{q}\) avec \(0\leq{p}\leq{1}\) Il permet de paramétrer le poids accordé au début où à la fin de temps d’observation. Si \(p=q=0\) on retrouve le test de base non pondéré1.
En pratique/remarques:
Les tests du log-rank sont sensibles à l’hypothèse de risques proportionnels (voir modèle semi-paramétrique de Cox). En pratique si des courbes de séjours se croisent, il est fortement déconseillé de les utiliser analystiquement. Cela ne signifie pas que si les courbes ne se croisent pas, l’hypothèse de proportionalité des risques est respectée : des rapports de risque peuvent au cours du temps s’intensifier, se réduire ou, le cas échant s’inverser, ce qui est typique d’un croisement.
Effectuer un test global (multiple/omnibus) sur un nombre important de groupes (ou >2) peut rendre les p-value artificiellement très basses (idem test classique d’indépendance). Il peut être intéressant de tester des courbes deux à deux (idem qu’une régression avec covariable discrète), en conservant un seul degré de liberté. Des méthodes de correction du test multiple sont possibles ou disponibles si on utilise R, bien que la bonne méthode n’a jamais fait consensus chez les statisticiens.
Note
Cette question de test multiple ne se pose pas dans le support. Mais c’est le cas avec la formation en live pour les données traitées dans les TP
On utilise la fonction survdiff de la librairie survival. Le résultat du test de Peto-Peto est affiché par défaut (rho=1). Si on souhaite utiliser le test non pondéré, on ajoute l’option rho=0. Pour obtenir le résultat d’un test multiple corrigé (plus d’un degré de liberté), on peut utiliser la fonction pairwise_survdiff du package survminer. Cette fonction permet également d’obtenir des tests 2 à 2.
Je conseille de rester sur l’option Peto-Peto et dans le cas d’une variable à plus de deux modalités, d’utiliser la fonction de survminerpairwise_survdiff.
On utilise la commande sts test avec le nom de la version du test: peto, wilcoxon . Sans préciser le nom de la variante, le test non pondéré est exécuté.
Avec la librairie lifelines, on utilise la fonction logrank_test. Quatre variantes sont disponibles (Wilcoxon, Tarone-Ware, Peto-Peto et Fleming-Harrigton). On peut également utiliser la fonction duration.survdiff de statmodels (non pondéré, Wilcoxon - appelé ici Breslow- et Tarone-Ware).
Le test non pondéré et la version Wilcoxon sont données avec l’option strata de la proc lifetest. Attention : ne jamais utiliser la version LR Test qui est biaisée. Pour obtenir d’autres versions du test du log-rank, on ajoute /test=all à l’option strata.
6.1.3 Application
On compare ici l’effet du pontage coronarien sur le risque de décéder depuis l’inscription dans le registre de greffe.
Résultats des tests du logrank
Test
df
Chi2
P>Chi2
Non pondéré
1
6.59
0.0103
Wilcoxon (Breslow)
1
8.99
0.0027
Tarone-Ware
1
8.46
0.0036
Peto-Peto
8.66
0.0033
Les résultats font apparaître que l’opération permet d’augmenter la durée de survie des personnes. Il apparait que la p-value est plus élevée pour test non pondérée. Cela peut-il s’expliquer en regardant les deux courbes de séjours? Qu’en est-il de la proportionalité des risques ???? …. Réponse pendant la formation.
6.2 Comparaison des RMST
RMST: Restricted Mean of Survival Time
La comparaison des RMST est une alternative pertinente aux tests du log-rank car elle ne repose pas sur des hypothèses contraignantes (proportionnalité des risques, distribution des censures), et permet une lecture vivante basée sur des espérances de séjour et non sur la lecture d’une simple p-value traduisant l’homogénéité ou non des fonctions de séjour. Par ailleurs les comparaisons sont souples, on peut choisir un ou plusieurs points d’horizon pour alimenter l’analyse.
Principe
L’aire sous la fonction de survie représente la durée moyenne d’attente jusqu’à l’évènement, soit une espérance de survie.
En présence de censure à droite, il faut borner la durée maximale \(t^*<\infty\). Cette espérance de survie ou de séjour s’interprète donc sur un horizon fini. On est très proche d’une mesure en analyse démographique type « espérance de vie partielle ».
\(RMST =\int_0^{t^*}S(t)dt\).
On peut facilement comparer les RMST de deux groupes, en termes de différence ou de ratio.
Par défaut on définit généralement \(t^*\) à partir le temps du dernier évènement observé. Il est néanmoins possible de calculer le RMST sur des intervalles plus court, ce qui lui permet une véritable souplesse au niveau de l’analyse.
R-Stata-Python-Sas
Attention, selon les logiciels la durée max par défaut n’est pas la même. Pour R et Sas, il s’agit du dernier évènement observé sur l’ensemble de l’échantillon, alors que Stata prend la durée qui correspond au dernier évènement observé le plus court des deux groupes . Cela affectera légèrement la valeur des Rmst estimées par défaut.
Pour l’exemple, la durée maximale utilisée par R est de 1407 jours alors que pour Stata elle est de 995 jours.
Librairie SurvRm2. Programmée par les mêmes personnes que la commande Stata, la fonction proposée n’est pas très souple malheureusement.
Commande externe strmst2. La plus ancienne fonction proposée par les logiciels. Au final plus limitée que la solution Sas. J’ai programmé une commande, diffrmst, qui représente graphiquement les estimations des Rmst pour chaque temps d’évènement, leurs différences et les p-value issues des comparaisons.
Estimation un peu pénible. A partir de l’estimateur KM obtenu avec la fonction KaplanMeierFitter de lifelines, on peut obtenir les RMST avec la fonction restricted_mean_survival_time. On peut tracer les fonctions, en revanche le test de comparaison n’est pas implémenté.
Disponible depuis la version 15.1 de SAS/Stat (fin 2018). Les estimations et le résultat du test de comparaison sont récupérables très simplement dans une proc lifetest, avec en option **plots=(rmst)** . Bien que sortie tardivement par rapport Stata et R, les résultats sont particulièrement complets.
Application
Avec \(tmax=1407\):
Estimation des Rmst pour la variable surgery
Groupes
RMST
Std. Err
95% CI
\(surgery=1\)
884.576
187.263
517.546 - 1251.605
\(surgery=0\)
379.148
61.667
258.282 - 500.014
Différences entre Rmst pour la variable surgery
Types de contraste
Ecarts RMST
P>|z|
95% CI
\(Rmst(surgery1 - surgery0)\)
505.428
0.010
517.546 - 1251.605
\(Rmst\left(\frac{surgery1}{surgery0}\right)\)
2.333
0.002
1.383 - 3.937
Ici \(t^*\) est égal à 1407 jours, soit la durée qui correspond au dernier décès observé.
Sur un horizon de 1407 jours, ces individus opérés d’un pontage peuvent espérer vivre 884 jours en moyenne, contre 379 jours pour les autres. La durée moyenne de survie est donc 2.3 fois plus importante pour les personnes opérées (rapport des Rmst = 2.3 ), ce qui correspond à une différence de 379 jours.
Le tableau et le graphique suivant donnent les valeurs des Rmst et les écarts de la variable surgery en faisant varier \(tmax\) sur chaque jour où au moins un décès a été observé. Il a été réalisé avec Stata, la durée maximale utilisée a été paramétrée à 1407 jours (idem R, Sas).
Comme le premier décès observé pour les personnes opéré se situe le 165eme jours, il est tout à fait normal que pour ce groupe de personnes la valeur de la Rmst soit identique au jour de décès des individus non opérés.
Note
Pour la version pdf, seulement une grosse dizaine de points a été sélectionné en raison de la longueur du tableau.
Comparaison des Rmst à chaque jour où au moins un décès est observé
Comparaison des Rmst à chaque jour où au moins un décès est observé
A ma connaissance il n’est pas implémenté dans R↩︎
Code source
---#title: "**Tests de comparaison**"---# Tests de comparaison- Les tests d'égalités des fonctions de survie entre différentes valeurs d'une covariable sont calculés à partir de la méthode de Kaplan Meier. - L'utilisation du test correspond à la nécessité de déterminer si une même distribution gouverne les évènements observés dans les différentes strates. - **Attention**: pas de test possible sur des variables quantitatives. Il faut donc prévoir des regroupements pour les transformer en variable ordinale. Deux méthodes sont utilisées: * La plus ancienne, la plus diffusée, et peut-être la moins bonne: test dits du **log-rank**). * Plus récente et (hélas) moins difusée: comparaison des **RMST** (*Restricted Mean of Survival Time*). ## Tests du log-rankIl s'agit d'une série de tests qui répondent à la même logique, la seule différence réside dans le poids accordé au début ou à la fin de la période d'observation. Par ailleurs ces différents tests sont plus ou moins sensibles à la distribution des censures à droites entre les sous échantillons et à l'hypothèses de proportionnalité des risques. Dans leur logique, ces tests entrent dans le cadre des tests d'indépendance du Khi2, même si formellement ils relèvent des techniques dites de **rang** (d'où le nom). Il s'agira donc de comparer des effectifs observés à des effectifs espérés à chaque moment d'évènement. La principale différence réside dans le calcul de la variance de la statistique du test qui, ici, suit assez logiquement une loi hypergéométrique [proche loi binomiale mais avec tirage avec remise]. ### Principe de calcul de la statistique de test* **Effectifs observés en $t_i$**: $o_{i1}$ et $o_{i2}$ sont égaux à $d_{i1}$ et $d_{i2}$, et leur somme pour tous les temps d'évènement à $O_1$ et $O_2$.* **Effectifs expérés** (hypothèse nulle $H_0$): comme pour une statistique du $\chi^2$ on se base sur les marges, avec le risque set ($R_i$) en $t_i$ pour dénombrer les effectifs, soit $e_{i1}=R_{i1}\times\frac{d_i}{R_i}$ et $e_{i2}=R_{i2}\times\frac{d_2}{R_i}$. Leur somme pour tous les temps d'évènement est égale à $E_1$ et $E_2$. Le principe de calcul des effectifs observés reposent donc sur l'hypothèse d'un rapport des risques toujours égal à 1 au cours du temps (*hypothèse fondamentale de risques proportionnels*).* **Statistique du log-rank**: $(O_1 - E_1) = -(O_2 - E_2)$. * **Statistique de test**: sous $H_0$, $\frac{(O_1 - E_1)^2}{\sum{v_i}}$, avec $v_i$ la variance de $(o_{i1} - e_{i2})$, suis un $\chi^2(1)$.Si on teste simultanément la différence de $g$ fonctions de survie, ce qui n'est pas une bonne idée en passant, la statistique de test suis un $\chi^2(g-1)$. ### Les principaux tests log-rankLe principe de construction des effectifs observés et espérés reste strictement le même dans chaque test, les différences résident dans les pondérations ($w_i$) qui prennent en compte, de manière différente, la taille de la population encore soumise au risque à chaque durée où au moins un évènement est observé. * **Test du log-rank**: $w_i=1$ Il accorde le même poids à toutes les durées d'évènement. C'est le test standard, le plus utilisé. Il n'y a donc pas de pondération au final.* **Test de Wilconxon-Breslow-Grehan**: $w_i=R_i$ Les écarts entre effectifs observés et espérés sont pondérés par la population soumise à risque en $t_i$. Le test accorde plus de poids au début de la période analysée, et il est sensible aux différences de distributions entre les strates des observations censurées.* **Test de Tarone-Ware**: $w_i=\sqrt{R_i}$ Variante du test précédent, il atténue le poids accordé aux évènements au début de la période d'observation. Il est par ailleurs moins sensible au problème de la distribution des censures entre les strates. * **Test de Peto-Peto** : $w_i=S_i$ La pondération est une variante de la fonction de survie KM (avec $R_i=R_i+1$). Le test n'est pas sensible au problème de distribution des censures. * **Test de Fleming-Harington**: $w_i=(S_i)^p\times(1-S_i)^{q}$ avec $0\leq{p}\leq{1}$ Il permet de paramétrer le poids accordé au début où à la fin de temps d'observation. Si $p=q=0$ on retrouve le test de base non pondéré^[A ma connaissance il n'est pas implémenté dans R]. **En pratique/remarques**: * Les tests du log-rank sont sensibles à l’hypothèse de risques proportionnels (voir **modèle semi-paramétrique de Cox**). En pratique si des courbes de séjours se croisent, il est fortement déconseillé de les utiliser analystiquement. Cela ne signifie pas que si les courbes ne se croisent pas, l’hypothèse de proportionalité des risques est respectée : des rapports de risque peuvent au cours du temps s'intensifier, se réduire ou, le cas échant s’inverser, ce qui est typique d’un croisement.* Effectuer un test global (multiple/omnibus) sur un nombre important de groupes (ou >2) peut rendre les p-value artificiellement très basses (idem test classique d'indépendance). Il peut être intéressant de tester des courbes deux à deux (idem qu’une régression avec covariable discrète), en conservant un seul degré de liberté. Des méthodes de correction du test multiple sont possibles ou disponibles si on utilise R, bien que la bonne méthode n'a jamais fait consensus chez les statisticiens. ::: callout-noteCette question de test multiple ne se pose pas dans le support. Mais c'est le cas avec la formation *en live* pour les données traitées dans les TP:::**R-Stata-Python-Sas** ::: panel-tabset#### R On utilise la fonction **`survdiff`** de la librairie `survival`. Le résultat du test de Peto-Peto est affiché par défaut (`rho=1`). Si on souhaite utiliser le test non pondéré, on ajoute l’option `rho=0`. Pour obtenir le résultat d'un test multiple corrigé (plus d’un degré de liberté), on peut utiliser la fonction **`pairwise_survdiff`** du package **`survminer`**. Cette fonction permet également d'obtenir des tests 2 à 2. Je conseille de rester sur l'option ***Peto-Peto*** et dans le cas d'une variable à plus de deux modalités, d'utiliser la fonction de `survminer` **`pairwise_survdiff`**.#### Stata On utilise la commande **`sts test`** avec le nom de la version du test: `peto`, `wilcoxon` . Sans préciser le nom de la variante, le test non pondéré est exécuté.#### PythonAvec la librairie `lifelines`, on utilise la fonction **`logrank_test`**. Quatre variantes sont disponibles (Wilcoxon, Tarone-Ware, Peto-Peto et Fleming-Harrigton). On peut également utiliser la fonction **`duration.survdiff`** de `statmodels` (non pondéré, Wilcoxon - appelé ici Breslow- et Tarone-Ware). #### Sas {{< fa solid cross>}}Le test non pondéré et la version Wilcoxon sont données avec l’option **`strata`** de la `proc lifetest`. Attention : ne jamais utiliser la version *LR Test* qui est biaisée. Pour obtenir d’autres versions du test du log-rank, on ajoute **`/test=all`** à l’option `strata`.:::### Application On compare ici l'effet du pontage coronarien sur le risque de décéder depuis l'inscription dans le registre de greffe. ::: {.box_img}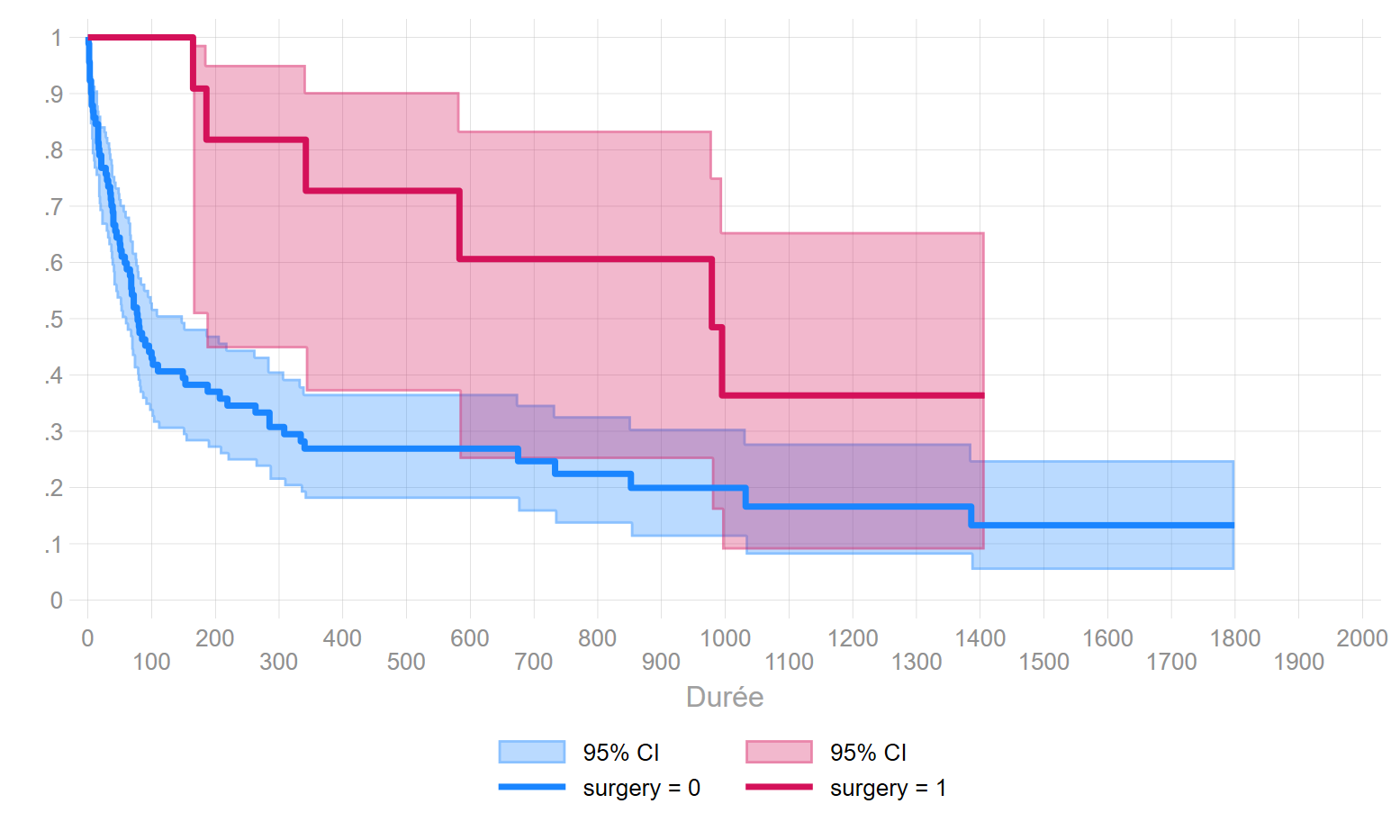{width=70%}:::| **Test** | **df** | **Chi2** | **P>Chi2** ||------------------------|--------|----------|-------------|| **Non pondéré** | 1 | 6.59 | 0.0103 || **Wilcoxon (Breslow**) | 1 | 8.99 | 0.0027 || **Tarone-Ware** | 1 | 8.46 | 0.0036 || **Peto-Peto** | | 8.66 | 0.0033 |: Résultats des tests du logrankLes résultats font apparaître que l’opération permet d’augmenter la durée de survie des personnes. Il apparait que la p-value est plus élevée pour test non pondérée. Cela peut-il s'expliquer en regardant les deux courbes de séjours? Qu'en est-il de la proportionalité des risques ???? .... Réponse pendant la formation.## Comparaison des RMST **RMST**: *Restricted Mean of Survival Time* La comparaison des RMST est une alternative pertinente aux tests du log-rank car elle ne repose pas sur des hypothèses contraignantes (proportionnalité des risques, distribution des censures), et permet une lecture vivante basée sur des espérances de séjour et non sur la lecture d’une simple p-value traduisant l’homogénéité ou non des fonctions de séjour. Par ailleurs les comparaisons sont souples, on peut choisir un ou plusieurs points d’horizon pour alimenter l’analyse. **Principe** * L’aire sous la fonction de survie représente la durée moyenne d’attente jusqu'à l’évènement, soit une espérance de survie.* En présence de censure à droite, il faut borner la durée maximale $t^*<\infty$. Cette espérance de survie ou de séjour s'interprète donc sur un horizon fini. On est très proche d’une mesure en analyse démographique type « espérance de vie partielle ».* $RMST =\int_0^{t^*}S(t)dt$. * On peut facilement comparer les RMST de deux groupes, en termes de différence ou de ratio. * Par défaut on définit généralement $t^*$ à partir le temps du dernier évènement observé. Il est néanmoins possible de calculer le RMST sur des intervalles plus court, ce qui lui permet une véritable souplesse au niveau de l’analyse. **R-Stata-Python-Sas** Attention, selon les logiciels la durée max par défaut n'est pas lamême. Pour R et Sas, il s'agit du dernier évènement observé sur l'ensemble de l'échantillon, alors que Stata prend la durée qui correspond au dernier évènement observé le plus court des deux groupes . Cela affectera légèrement la valeur des Rmst estimées par défaut. Pour l'exemple, la durée maximale utilisée par R est de 1407 jours alors que pour Stata elle est de 995 jours.::: panel-tabset#### R Librairie **`SurvRm2`**. Programmée par les mêmes personnes que la commande Stata, la fonction proposée n'est pas très souple malheureusement.#### StataCommande externe **`strmst2`**. La plus ancienne fonction proposée par les logiciels. Au final plus limitée que la solution Sas. J’ai programmé une commande, `diffrmst`, qui représente graphiquement les estimations des Rmst pour chaque temps d’évènement, leurs différences et les p-value issues des comparaisons. #### PythonEstimation un peu pénible. A partir de l'estimateur KM obtenu avec la fonction `KaplanMeierFitter` de `lifelines`, on peut obtenir les RMST avec la fonction `restricted_mean_survival_time`. On peut tracer les fonctions, en revanche le test de comparaison n'est pas implémenté.#### SAS {{< fa solid cross>}} Disponible depuis la version 15.1 de SAS/Stat (fin 2018). Les estimations et le résultat du test de comparaison sont récupérables très simplement dans une `proc lifetest`, avec en option **`plots=(rmst)**` . Bien que sortie tardivement par rapport Stata et R, les résultats sont particulièrement complets.:::**Application** Avec $tmax=1407$:| Groupes | RMST | Std. Err | 95% CI ||-------------|---------|----------|--------------------|| $surgery=1$ | 884.576 | 187.263 | 517.546 - 1251.605 || $surgery=0$ | 379.148 | 61.667 | 258.282 - 500.014 |: Estimation des Rmst pour la variable surgery| Types de contraste | Ecarts RMST | P\>\|z\| | 95% CI ||----------------------------------------------|--------------|-----------|--------------------|| $Rmst(surgery1 - surgery0)$ | 505.428 | 0.010 | 517.546 - 1251.605 || $Rmst\left(\frac{surgery1}{surgery0}\right)$ | 2.333 | 0.002 | 1.383 - 3.937 |: Différences entre Rmst pour la variable surgeryIci $t^*$ est égal à 1407 jours, soit la durée qui correspond au dernier décès observé. Sur un horizon de 1407 jours, ces individus opérés d'un pontage peuvent espérer vivre 884 jours en moyenne, contre 379 jours pour les autres. La durée moyenne de survie est donc 2.3 fois plus importante pour les personnes opérées (rapport des Rmst = 2.3 ), ce qui correspond à une différence de 379 jours.Le tableau et le graphique suivant donnent les valeurs des Rmst et les écarts de la variable *surgery* en faisant varier $tmax$ sur chaque jour où au moins un décès a été observé. Il a été réalisé avec Stata, la durée maximale utilisée a été paramétrée à 1407 jours (idem R, Sas).Comme le premier décès observé pour les personnes opéré se situe le 165eme jours, il est tout à fait normal que pour ce groupe de personnes la valeur de la Rmst soit identique au jour de décès des individus non opérés.::: callout-notePour la version pdf, seulement une grosse dizaine de points a été sélectionné en raison de la longueur du tableau.:::::: {.content-visible when-format="html"}```{r eval=FALSE}#| code-fold: true#| code-summary: "**Afficher le tableau**" +--------------------------------------------------------------------------+ | _time _rmst1 _rmst0 _diff 95%CI lower 95%CI upper pvalue| |--------------------------------------------------------------------------| |--------------------------------------------------------------------------| | 1 1 1 0 0 0 . | | 2 2 1.989011 .010989 -.0104304 .0324084 .3146368 | | 3 3 2.945055 .0549451 -.0009099 .1108 .0538507 | | 5 5 4.791209 .2087912 .0549289 .3626535 .0078217 | | 6 6 5.692307 .3076923 .0995576 .5158269 .0037617 | |--------------------------------------------------------------------------| | 8 8 7.45055 .5494505 .2224352 .8764658 .0009908 | | 9 9 8.318682 .6813186 .2913915 1.071246 .0006156 | | 11 11 10.03297 .9670329 .4461406 1.487925 .0002741 | | 12 12 10.89011 1.10989 .5212656 1.698514 .0002193 | | 16 16 14.27415 1.725846 .8588567 2.592834 .0000956 | |--------------------------------------------------------------------------| | 17 17 15.08677 1.91323 .9769751 2.849484 .000062 | | 18 18 15.88825 2.111745 1.10533 3.118161 .0000391 | | 21 21 18.25931 2.740688 1.516228 3.965148 .0000115 | | 28 28 23.63593 4.364065 2.598741 6.129389 1.26e-06 | | 30 30 25.14985 4.85015 2.924714 6.775586 7.93e-07 | |--------------------------------------------------------------------------| | 31 31 25.89568 5.104324 3.098862 7.109787 6.08e-07 | | 32 32 26.6415 5.358499 3.272218 7.444779 4.80e-07 | | 35 35 28.84508 6.154923 3.825102 8.484744 2.24e-07 | | 36 36 29.5683 6.431698 4.020457 8.842939 1.71e-07 | | 37 37 30.28023 6.719774 4.227545 9.212004 1.26e-07 | |--------------------------------------------------------------------------| | 39 39 31.68147 7.318526 4.664121 9.972931 6.52e-08 | | 40 40 32.3708 7.629202 4.893653 10.36475 4.60e-08 | | 43 43 34.37097 8.629034 5.651719 11.60635 1.34e-08 | | 45 45 35.68181 9.318189 6.177435 12.45894 6.07e-09 | | 50 50 38.90242 11.09758 7.539261 14.6559 9.80e-10 | |--------------------------------------------------------------------------| | 51 51 39.53524 11.46476 7.822028 15.10748 6.89e-10 | | 53 53 40.77829 12.22171 8.410756 16.03267 3.27e-10 | | 58 58 43.82939 14.17061 9.933216 18.408 5.58e-11 | | 61 61 45.62615 15.37385 10.87721 19.87049 2.07e-11 | | 66 66 48.56425 17.43575 12.50275 22.36874 4.28e-12 | |--------------------------------------------------------------------------| | 68 68 49.71689 18.28311 13.175 23.39121 2.30e-12 | | 69 69 50.27061 18.72939 13.53629 23.92248 1.56e-12 | | 72 72 51.89787 20.10213 14.65526 25.54901 4.71e-13 | | 77 77 54.49696 22.50304 16.63758 28.36851 5.51e-14 | | 78 78 55.00547 22.99453 17.04528 28.94377 3.57e-14 | |--------------------------------------------------------------------------| | 80 80 55.99991 24.00009 17.88509 30.11509 1.44e-14 | | 81 81 56.48582 24.51418 18.31722 30.71113 8.88e-15 | | 85 85 58.38429 26.61571 20.09202 33.1394 1.33e-15 | | 90 90 60.70087 29.29913 22.36311 36.23515 2.22e-16 | | 96 96 63.41296 32.58704 25.15034 40.02373 0 | |--------------------------------------------------------------------------| | 100 100 65.17583 34.82418 27.05229 42.59606 0 | | 102 102 66.03465 35.96535 28.0274 43.90329 0 | | 109 109 68.96146 40.03855 31.5203 48.55679 0 | | 110 110 69.37957 40.62043 32.0176 49.22326 0 | | 131 131 77.91607 53.08393 42.67269 63.49517 0 | |--------------------------------------------------------------------------| | 149 149 85.23307 63.76693 51.71735 75.81651 0 | | 153 153 86.81125 66.18875 53.7781 78.5994 0 | | 165 165 91.40231 73.59769 60.11884 87.07655 0 | | 180 178.6364 97.14113 81.49523 66.43188 96.55859 0 | | 186 184.0909 99.43666 84.65425 68.8452 100.4633 0 | |--------------------------------------------------------------------------| | 188 185.7273 100.2018 85.52544 69.46069 101.5902 0 | | 207 201.2727 107.2365 94.0362 75.10156 112.9708 0 | | 219 211.0909 111.5314 99.55952 78.49335 120.6257 0 | | 263 247.0909 126.7362 120.3547 90.2882 150.4212 4.22e-15 | | 265 248.7273 127.4026 121.3246 90.82352 151.8258 6.44e-15 | |--------------------------------------------------------------------------| | 285 265.0909 134.0671 131.0238 96.0849 165.9628 1.98e-13 | | 308 283.9091 141.1416 142.7675 102.6688 182.8662 2.99e-12 | | 334 305.1818 148.8057 156.3761 110.3439 202.4082 2.77e-11 | | 340 310.0909 150.4975 159.5934 112.1866 207.0003 4.16e-11 | | 342 311.7273 151.0358 160.6915 112.8294 208.5536 4.69e-11 | |--------------------------------------------------------------------------| | 370 332.0909 158.5717 173.5192 119.5852 227.4532 2.87e-10 | | 397 351.7273 165.8385 185.8887 125.7134 246.0641 1.41e-09 | | 427 373.5454 173.9127 199.6327 132.2074 267.058 6.51e-09 | | 445 386.6364 178.7573 207.8791 135.9845 279.7736 1.45e-08 | | 482 413.5454 188.7155 224.83 143.5408 306.1191 5.93e-08 | |--------------------------------------------------------------------------| | 515 437.5454 197.5971 239.9483 150.1031 329.7935 1.65e-07 | | 545 459.3636 205.6714 253.6923 155.9623 351.4222 3.62e-07 | | 583 487 215.8987 271.1013 163.2743 378.9283 8.32e-07 | | 596 494.8788 219.3976 275.4812 164.6331 386.3293 1.11e-06 | | 620 509.4243 225.8569 283.5673 166.951 400.1836 1.88e-06 | |--------------------------------------------------------------------------| | 670 539.7273 239.314 300.4133 171.1152 429.7114 5.27e-06 | | 675 542.7576 240.6597 302.0979 171.4897 432.7061 5.80e-06 | | 733 577.9091 254.969 322.9401 176.861 469.0191 .0000147 | | 841 643.3636 279.1917 364.1719 187.8796 540.4643 .0000515 | | 852 650.0303 281.6588 368.3715 188.9058 547.8372 .0000575 | |--------------------------------------------------------------------------| | 915 688.2121 294.2187 393.9934 196.3119 591.6749 .0000937 | | 941 703.9697 299.4022 404.5675 199.2493 609.8857 .0001125 | | 979 727 306.978 420.022 203.4389 636.6051 .0001441 | | 995 734.7576 310.1678 424.5898 204.0643 645.1152 .0001609 | | 1032 748.2121 317.5443 430.6678 202.7468 658.5889 .0002127 | |--------------------------------------------------------------------------| | 1141 787.8485 335.6531 452.1953 200.7097 703.681 .0004248 | | 1321 853.303 365.5577 487.7454 191.5434 783.9473 .0012492 | | 1386 876.9394 376.3565 500.5829 186.9499 814.2158 .0017585 | | 1400 882.0303 378.2173 503.813 186.4392 821.1869 .0018625 | | 1407 884.5757 379.1476 505.4281 186.1745 824.6817 .0019162 | +--------------------------------------------------------------------------+```:::::: {.content-visible when-format="pdf"}```{r eval=FALSE}# Seulement une partie de l'output: se reporter à la version html +--------------------------------------------------------------------------+ | _time _rmst1 _rmst0 _diff 95%CI lower 95%CI upper pvalue| |--------------------------------------------------------------------------| | 1 1 1 0 0 0 . | | 2 2 1.989011 .010989 -.0104304 .0324084 .3146368 | | 3 3 2.945055 .0549451 -.0009099 .1108 .0538507 | | 5 5 4.791209 .2087912 .0549289 .3626535 .0078217 | | 6 6 5.692307 .3076923 .0995576 .5158269 .0037617 | |--------------------------------------------------------------------------| | 8 8 7.45055 .5494505 .2224352 .8764658 .0009908 | | 9 9 8.318682 .6813186 .2913915 1.071246 .0006156 | | 50 50 38.90242 11.09758 7.539261 14.6559 9.80e-10 | | 515 437.5454 197.5971 239.9483 150.1031 329.7935 1.65e-07 | | 995 734.7576 310.1678 424.5898 204.0643 645.1152 .0001609 | | 1032 748.2121 317.5443 430.6678 202.7468 658.5889 .0002127 | |--------------------------------------------------------------------------| | 1141 787.8485 335.6531 452.1953 200.7097 703.681 .0004248 | | 1321 853.303 365.5577 487.7454 191.5434 783.9473 .0012492 | | 1386 876.9394 376.3565 500.5829 186.9499 814.2158 .0017585 | | 1400 882.0303 378.2173 503.813 186.4392 821.1869 .0018625 | | 1407 884.5757 379.1476 505.4281 186.1745 824.6817 .0019162 | +--------------------------------------------------------------------------+```:::::: {.box_img}{width=70%}:::